8 déc. 22

Temps de lecture estimé :min
Biodiversité, maladies émergentes, épidémies… Révéler l’invisible grâce à l’ADN présent dans l’environnement
Marine Combe, Institut de recherche pour le développement (IRD) et Rodolphe Gozlan, Institut de recherche pour le développement (IRD)Nous n’en avons pas conscience, mais nous laissons tous notre empreinte génétique dans les lieux que nous fréquentons. En effet, sans nous en apercevoir, nous disséminons quotidiennement notre ADN autour de nous. Il est présent non seulement dans les cellules que nous perdons quotidiennement durant les processus de renouvellement cellulaire, mais aussi dans nos sécrétions (telles que la salive, le mucus) ou nos excrétions (les urines ou les fèces)… Et cela se poursuit après notre mort, puisque nos cadavres en décomposition relarguent eux aussi de l’ADN.
Nous ne sommes pas les seuls à laisser de telles traces de notre passage : c’est également le cas de tous les organismes vivants, des bactéries aux parasites en passant par les plantes, les champignons ou encore les virus. Or, une fois l’ADN libéré dans l’environnement, il peut être prélevé et analysé grâce à la biologie moléculaire et aux nouvelles technologies de séquençage.
Si les séries télévisées et certains procès marquants ont popularisé l’utilisation de ces techniques par la police scientifique, l’intérêt d’étudier les molécules d’ADN présentes dans l’environnement ne s’arrête pas là.
Saviez-vous, par exemple, que leur analyse peut nous permettre de mieux anticiper l’émergence de nouvelles maladies infectieuses ? De surveiller activement l’évolution d’une épidémie en cours ? De suivre l’évolution de la biodiversité ? Explications.
Qu’est-ce que l’ADN environnemental, et comment l’analyse-t-on ?
L’ADN environnemental (ADNe) correspond à l’ensemble des molécules d’ADN présentes dans un milieu donné. Il s’agit de l’ADN qui a été relâché dans leur milieu par les espèces qui y vivent. Sa collecte se fait en prélevant des échantillons de diverses natures : eau douce, saumâtre ou marine, eaux usées, sédiments, sol, fèces, air ou encore glace.
Une fois les échantillons récupérés, l’ADN en est extrait. On obtient alors une sorte de « soupe d’ADN », dans laquelle on va aller « pêcher » des séquences d’ADN spécifiques de certains types d’organismes en les amplifiant grâce à la technique de la PCR (polymerase chain reaction ou « réaction de polymérisation en chaîne »). On compare ensuite les résultats obtenus avec les données contenues dans des bases de données de séquences d’ADN, afin de déterminer précisément à quelles espèces appartiennent les ADN amplifiés.
À partir de l’ADN relâché par les espèces dans leur environnement, on peut donc rapidement identifier non seulement leur présence, mais aussi avoir une idée de leur quantité, en pratiquant une PCR quantitative en temps réel, ou qPCR. Cette méthode fait correspondre le nombre de cycles d’amplification de l’ADN avec une concentration, grâce à l’utilisation d’un échantillon d’ADN « contrôle », de concentration connue.
Il faut cependant savoir que la préservation de l’ADN dans l’environnement dépend des conditions dans lesquelles il se trouve. L’ADN est notamment une molécule très sensible à la température : il sera donc mieux conservé aux basses températures. En outre, dans l’environnement l’ADN se fragmente aussi à cause de la présence de nucléases (des enzymes capables de découper l’ADN). Ce n’est toutefois pas forcément un problème, car les analyses d’ADNe ont justement pour objectif de cibler de courts fragments d’ADN.
Mesurer les conséquences des changements environnementaux
Les analyses d’ADNe présentent plusieurs avantages techniques.
Non invasive, rapide, et relativement peu coûteuse, cette approche permet d’obtenir une « photographie » de la biodiversité d’un lieu à un moment donné. On peut répéter l’opération à différents intervalles de temps, de façon à voir son évolution. Il est ainsi possible de connaître l’état de la biodiversité sur de larges échelles spatiales et temporelles.
Il est de cette façon possible de suivre rapidement et régulièrement l’impact de certains changements environnementaux sur les espèces qui peuplent les écosystèmes.
C’est par ailleurs une méthode qui peut être facilement transférée aux acteurs locaux, à des volontaires non scientifiques et qui peuvent être déployées dans les pays à haut comme à faibles revenus.
L’avantage est aussi important par rapport aux méthodes « classiques », qui reposent sur le prélèvement puis l’identification d’un maximum d’espèces vivant au sein d’un milieu donné.
L’étape de collecte est en effet une tâche souvent longue et parfois fastidieuse. Qui plus est, cela nécessite que soient disponibles des spécialistes capables de caractériser tous les compartiments d’un écosystème, des bactéries aux petits mammifères, en passant par les insectes, les végétaux, etc. Dans les forêts tropicales, où la biodiversité est à son apogée (et où notre connaissance des espèces reste très limitée), il s’agit d’un travail titanesque. L’analyse d’ADNe permet de s’affranchir de ces contraintes.
Le cas des bactéries est très parlant. Auparavant, pour savoir lesquelles étaient présentes dans un échantillon, il fallait d’abord préparer des cultures à partir dudit échantillon, puis identifier dans un second temps les colonies formées. Le problème est que les conditions de culture de la majorité des bactéries sont inconnues : un grand nombre d’entre elles ne peuvent donc être détectées de cette façon. Grâce à l’ADNe, plus besoin de passer par cette étape de culture, puisque la méthode permet de cibler spécifiquement des séquences d’ADN contenues dans un mélange complexe.
De nombreuses applications
L’ADNe est largement utilisé pour caractériser la nature dans son ensemble. Son analyse permet non seulement d’appréhender la biodiversité des plantes, arthropodes et autres champignons, mais aussi celle des communautés microbiennes, et ce au sein d’environnements très variés tels que le sol, les eaux douces, les eaux de mer, et même le corps humain.
Les analyses d’ADNe sont mises en œuvre dans des domaines aussi variés que l’écologie et la conservation des écosystèmes, la détection d’espèces invasives ou d’espèces rares, ou les études de paléogénétique (étude génétique des organismes et populations du passé grâce à de l’ADN ancien).
Mais ce n’est pas tout : elles sont aussi couramment utilisées pour effectuer de la biosurveillance et étudier les pollutions environnementales (pour évaluer par exemple l’état d’un cours d’eau). Et plus récemment, une nouvelle utilisation de l’ADNe a vu le jour, avec des résultats très prometteurs : la surveillance de l’émergence de pathogènes responsables de maladies infectieuses touchant les plantes, les animaux ou l’être humain.
L’ADNe au service de la détection des maladies infectieuses émergentes
De nombreuses maladies zoonotiques (transmises à l’être humain à partir des animaux) ont un lien direct ou indirect avec les milieux aquatiques. Les écosystèmes aquatiques constituent des accumulateurs d’ADN d’origines très variées. On y trouve bien entendu des ADN provenant d’organismes aquatiques, mais aussi ceux charriés par les cours d’eau, ceux qui proviennent de la terre (suite à l’érosion), ceux qui sont amenés par les aérosols, ou par les êtres vivants qui vivent aux abords desdits milieux aquatiques ou les fréquentent.
Deux types d’études peuvent être effectuées via les analyses d’ADNe. Il est tout d’abord possible de suivre l’augmentation de la charge en organismes pathogènes dans l’environnement naturel, et ainsi d’identifier des zones et des périodes à risque d’émergence d’une maladie infectieuse. Des travaux menés par l’Institut de recherche pour le développement (IRD) sur l’ulcère de Buruli (une maladie bactérienne provoquée par un microbe de la famille de la tuberculose et de la lèpre qui provoque des nécroses de la peau) et la leptospirose (une maladie bactérienne véhiculée par les rongeurs, pouvant entraîner une grave insuffisance rénale) en Guyane française a ainsi permis de mettre en évidence la distribution spatiale et saisonnière de ces agents infectieux zoonotiques dans les eaux douces et dans les sédiments.
Une seconde approche consiste à étudier les eaux usées des villes et des villages. S’y retrouvent en effet les pathogènes excrétés par les personnes infectées. En surveillant leur quantité, on peut détecter une émergence en cours au sein de la population. La comparaison des signaux infectieux à la sortie des villes, par rapport au bruit de fond « naturel » dans les écosystèmes aquatiques, peut aider à mieux comprendre les dynamiques infectieuses, et à identifier non seulement des foyers d’infection, mais aussi à déterminer leur saisonnalité.
Les analyses d’ADNe dans les eaux usées sont largement utilisées à travers le monde pour surveiller les émergences et les ré-émergences de maladies infectieuses humaines entériques, comme les norovirus, responsables de gastro-entérites, les virus de l’hépatite A, les virus influenza et les poliovirus.
L’analyse des ADNs présents dans les eaux usées a notamment largement été déployée au début de la pandémie de Covid-19 afin de détecter le virus SARS-CoV-2 dans les eaux usées en Chine, en Australie, aux Pays-Bas, aux États-Unis, en Espagne ou encore en France, par exemple. En Guyane française, notre équipe a utilisé ces approches de façon à détecter et suivre la circulation du virus dans les eaux usées, mais aussi de suivre l’évolution des variants du SARS-CoV-2 au cours du temps.
Cette surveillance en ADNe du Covid-19 dans les eaux usées permettait d’avoir environ deux semaines d’avance sur l’émergence de l’épidémie dans la population locale.
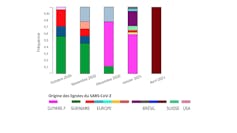
De telles données, récoltées de façon régulière selon un maillage prédéfini autour des villes, pourraient servir de support décisionnel à des préfectures et des Agences Régionale de Santé, en concertation avec les hôpitaux locaux, pour mieux anticiper les crises épidémiques.
Une approche de ce type est déclinable rapidement dans n’importe quelle partie du monde, car il suffit de prélever de l’eau ou des sédiments. Par ailleurs, plus le nombre d’études réalisées s’accroît, plus les bases de données de référence grandissent, ce qui facilite ensuite les analyses.
La détection d’un pathogène n’est pas synonyme de risque infectieux
Un point important à souligner en ce qui concerne l’étude des maladies infectieuses émergentes grâce à l’ADNe est que la détection de l’ADN d’un pathogène dans un environnement donné et à un moment donné ne peut à elle seule être interprétée en termes de risque infectieux.
En effet, il est important de garder à l’esprit que l’ADN d’un pathogène reste inchangé, quel que soit le statut biologique du pathogène : vivant, mort, infectieux, non infectieux. C’est aussi le cas si le pathogène a été détruit et que son ADN a été libéré dans l’environnement. Détecter l’ADN d’un pathogène dans un échantillon ne permet donc pas de conclure sur son statut infectieux.
Pour cette raison, il est nécessaire d’effectuer un suivi de la variation du nombre d’échantillons positifs comparé à l’ensemble des échantillons analysés, ainsi que de la concentration (charge) en ADN. Ces suivis de prévalence et de charge au cours du temps pourront renseigner sur une potentielle émergence.
Concernant l’estimation du risque infectieux, il faut, pour une pathogène donnée, savoir s’il s’agit bien d’une souche ou d’un variant infectieux (certaines bactéries Escherichia coli sont par exemple inoffensives, alors que d’autres fabriquent de dangereuses toxines, les shiga-toxines, responsables de diarrhées souvent sanglantes). Cette différenciation peut se faire par exemple en ciblant des gènes de virulence connus chez les souches/variants infectieux.
Comparer le signal environnemental avec la répartition spatiale et temporelle des cas cliniques de maladies, passés et présents, est également indispensable lors des analyses de risque infectieux via l’ADNe. Il s’agit d’analyser conjointement les données écologiques, microbiologiques et cliniques (médicales), car ces dernières sont absolument cruciales dans l’interprétation du risque infectieux environnemental ou d’émergence d’une maladie au sein d’une population.
ADN environnemental : une approche prometteuse malgré des limitations
Bien que très prometteuses, les approches d’ADNe sont aussi confrontées à plusieurs biais méthodologiques.
Tout d’abord, l’intensité du signal d’ADNe dépend de la disponibilité de l’ADN et de sa stabilité. Dans certains environnements, celui-ci est trop dégradé pour pouvoir être identifié (faux négatifs). Des inhibiteurs enzymatiques peuvent aussi être présents dans l’échantillon et bloquer l’amplification de l’ADN, qui n’est alors pas détecté (faux négatifs). À l’inverse, la contamination des échantillons peut être à l’origine de la détection de faux positifs. Parfois, également, les amplifications de l’ADN ciblé ne sont pas suffisamment spécifiques et amplifient aussi d’autres ADNs, non-cibles (faux positifs). Enfin, les bases de données de génomes de référence peuvent être manquantes ou incomplètes.
Malgré ces limitations, les analyses d’ADNe constituent un puissant outil sentinelle, qui permet de mesurer rapidement et de façon non invasive les changements de biodiversité qui s’opèrent dans la nature qui nous entoure, qu’ils soient microscopiques ou macroscopiques, et qu’ils concernent la faune, la flore ou les microorganismes. Elles sont aussi très prometteuses, quoiqu’encore exploratoires, en ce qui concerne l’étude des émergences de maladies infectieuses.
En Europe, les agences environnementales ont déjà mis en place des programmes d’échantillonnages annuels et saisonniers. C’est aussi le cas de nombreux pays hors Union européenne. Le déploiement de ces approches est notamment au cœur du projet européen BCOMING, mais aussi de l’initiative PREZODE.
Ces suivis de la circulation de pathogènes zoonotiques grâce à l’ADNe s’inscrivent dans l’approche « One Health » (« Une Seule Santé », celle des personnes, des animaux et des écosystèmes). Ils permettront d’obtenir une vision plus globale de la diversité des pathogènes qui nous entourent, mais aussi de mieux anticiper les risques et les foyers d’émergences des maladies infectieuses zoonotiques.![]()
Marine Combe, Chargée de recherche, Institut de recherche pour le développement (IRD) et Rodolphe Gozlan, Directeur de recherche, Institut de recherche pour le développement (IRD)
Nous remercions les auteurs et The Conversation pour l'autorisation de republication.
